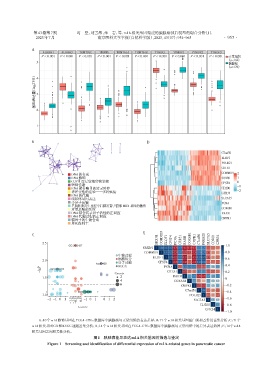
Page 71 - 南京医科大学学报自然科学版
P. 71
第43卷第7期 时 坚,刘雪昂,朱 岩,等. m1A相关基因构建的胰腺癌预后模型的综合分析[J].
2023年7月 南京医科大学学报(自然科学版),2023,43(07):954-965 ·957 ·
A
ALKBH1 ALKBH3 TRMT10C TRMT6 TRMT61A TRMT61B YTHDC1 YTHDF1 YTHDF2 YTHDF3
P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 P < 0.001 正常组织
(n=162)
5 胰腺癌
(n=178)
( log2TPM ) 4
基因表达量 3
2
1
B D
C7orf50
ELOF1
POLR2I
CRLS1
DNA的合成 COMMD9 2
RNA修饰 SRXN1 1
COPⅡ涂层囊泡货物装载 CPSF4 0
嘧啶代谢 -1
RNA聚合酶Ⅱ的转录延伸 CD200
神经变性的途径——多种疾病 GPR34 -2
RNA的代谢
线粒体基因表达 SLC7A5
小分子运输 PCNA
早期胚胎发生过程中后脑发育中前部 HOX 基因的激活
对氨基酸的反应 COX6B1
DNA结合转录因子活性的正调控 RARB
DNA代谢过程的正调控
辅因子的生物合成 CMPK1
坏死性凋亡
E
C
SRXN1 COMMD9 ELOF1 CPSF4 PCNA CRLS1 RARB COX6B1 CMPK1 C7orf50 POLR2I SLC7A5 CD200 GPR34
2.5
1.0
SRXN1 1 0.86 0.88 0.87-0.79 0.79 -0.8-0.79-0.760.73 0.75-0.68-0.58-0.56
COMMD9 1 0.88 0.83-0.79 0.77-0.75 -0.7-0.78 0.78 0.79-0.71-0.51-0.49 0.8
生物过程
细胞组分 ELOF1 1 0.89-0.82 0.77-0.83-0.63-0.810.83 0.89-0.72-0.52-0.61 0.6
2.0
分子功能 CPSF4 1 -0.78 0.77-0.83-0.65-0.8 0.83 0.84-0.66-0.61-0.66
KEGG PCNA 1 -0.65 0.75 0.76 0.75-0.71-0.69 0.64 0.44 0.58 0.4
-lgP
Counts CRLS1 1 -0.73-0.6-0.58 0.72 0.69-0.69-0.66-0.6 0.2
1.5 2 RARB 1 0.61 0.72-0.78-0.790.65 0.54 0.65 0
4 COX6B1 1 0.63-0.52-0.390.57 0.38 0.33
6 -0.2
CMPK1 1 -0.72-0.760.54 0.36 0.5
C7orf50 1 0.84-0.62-0.48-0.62
-0.4
1.0 POLR2I 1 -0.64-0.5-0.64
-1.5 -1.0 -0.5
-2 -1 0 1 0 0.5 -1 0 1 0 1 2 SLC7A5 1 0.46 0.37 -0.6
CD200 1 0.56 -0.8
z⁃score
GPR34 1
-1.0
A:10个m1A修饰基因在TCGA⁃GTEx数据库中胰腺癌与正常组织的表达差异;B:71个m1A相关基因蛋白质相互作用富集分析;C:71个
m1A相关基因GO和KEGG通路富集分析;D:14个m1A相关基因在TCGA⁃GTEx数据库中胰腺癌与正常组织中的差异表达热图;E:14个m1A
相关基因之间相关性分析。
图1 胰腺癌差异表达m1A相关基因的筛选与鉴定
Figure 1 Screening and identification of differential expression of m1A⁃related genes in pancreatic cancer