Page 28 - 南京医科大学学报自然科学版
P. 28
第42卷第8期
·1070 · 南 京 医 科 大 学 学 报 2022年8月
(papA、papC、papD)、编码 CFA/I 菌毛的基因(cfaA、 Stc(Salmonella)⁃STC E. coli str.
Peg(Salmonella)⁃Peg K⁃12 substr.
cfaB、cfaC、cafD/E)、编码自主转运蛋白的基因(aa⁃ AdeFGH efflux pump/transport autoinducer MG1655
Flagella(cluster Ⅰ)(Yersinia) J2_5
tA、cah、cdiA)、编 码 脑 内 皮 细 胞 侵 袭 基 因(ibeB、 ACET6SS
ibeC)、编码TTSS效应器分泌系统基因(espL1、espL4、 EspX5
EspX4
espR1、espX1、espX4、espX5)、编码 ACE T6SS 效应器 EspX1
EspR1
分泌系统基因aec15、编码类似鼠疫耶尔森菌鞭毛的 EspL4
EspL1
fliC基因、编码与生物膜形成有关的adeG、及编码菌 Invasion of brain endothelial cells(Ibes)
毛黏附决定因子的pegB、stcC等。其中,adeG、pegB、 Contact⁃dependentinhibition CDI system
Cah
stcC 3 个编码基因为 J2_5 菌株独有,在其他大肠杆 Antigen 43
AatA
菌中(如 E.coli O157:H7 str. EDL933、E.coli APEC Type Ⅰ fimbriae
P fimbriae
O1、E.coli CFT073等21株大肠杆菌)均未发现。 Hemorrhagic E. coli pilus(HCP)
2.7 原噬菌体的预测及验证 E. coli laminin⁃binding fimbriae(ELF)
E. coli common pilus(ECP)
本研究使用 Prophage Hunter 噬菌体预测工具 CFA/I fimbriae
0 2 4 6 8 10
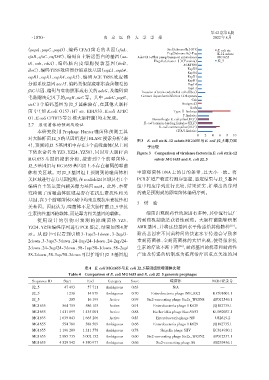
对大肠杆菌J2_5的基因组进行BLAST搜索分析(表 图 3 E. coli str.K⁃12 substr.MG1655 与 E. coli J2_5 毒力因
4),预测到J2_5基因组中存在3个前噬菌体(从上到 子比较
下依次命名为 YZ3、YZ24、YZ58),同时对大肠杆菌 Figure 3 Comparison of virulence factors in E. coli str.k⁃12
MG1655 基因组搜索分析,搜索到 7 个前噬菌体。 substr.MG1655 and E. coli J2_5
J2_5基因组与MG1655基因组上不存在相同的噬菌
体相关区域。对 J2_5 基因组上预测到的噬菌体相 中原噬菌体 DNA 上的目的条带,且大小一致。将
关区域进行毒力基因检测,在scaffold24区域只有1个 PCR扩增产物进行测序鉴定,鉴定结果与J2_5基因
编码自主转运蛋白相关毒力基因 aatA。此外,本研 组中相应序列进行比较,结果证实,扩增出的序列
究检测了前噬菌体区域是否存在抗生素抗性相关 的确是预测到的原噬菌体编码序列。
基因,在3个前噬菌体区域中均未发现抗生素抗性相
3 讨 论
关基因。因此认为,噬菌体不是大肠杆菌J2_5中抗
生素抗性基因的载体,而是毒力相关基因的载体。 细菌出现耐药性的原因有多种,其中流行最广
使 用 设 计 的 引 物 对 预 测 的 原 噬 菌 体 YZ3、 的耐药机制就是获得性耐药。大肠杆菌能够积累
YZ24、YZ58 编码序列进行 PCR 鉴定,结果如图 4 所 AMR 基因,并将这些基因水平传递给其他物种 [19] 。
示。从图中可以看到引物 3⁃1up/3⁃1down、3⁃2up/3⁃ 耐药基因在不同菌种间的快速水平传递会导致多
2down、3⁃3up/3⁃3down、24⁃1up/24⁃1down、24⁃2up/24⁃ 重耐药菌株、全耐药菌株的大量出现,使得很多抗
2down、24⁃3up/24⁃3down、58⁃1up/58⁃1down、58⁃2up/ 生素的疗效不断下降 [20] ,耐药基因的携带和耐药性
58⁃2down、58⁃3up/58⁃3down可以扩增出J2_5基因组 产生及传递的机制成为临床治疗所重点关注的问
表4 E. coli MG1655与E. coli J2_5基因组原噬菌体比较
Table 4 Comparison of E. coli MG1655 and E. coli J2_5 genomic prophages
Sequence ID Start End Category Score 噬菌体 NCBI登录号
J2_5 47 493 57 711 Ambiguous 0.63 N/A —
J2_5 1 236 14 979 Ambiguous 0.70 Enterobacteria phage IME_EC2 KF591601.1
J2_5 205 16 199 Active 0.99 Stx2⁃converting phage Stx2a_WGPS8 AP012540.1
MG1655 564 755 586 103 Active 0.91 Enterobacteria phage HK629 JQ182735.1
MG1655 1 411 899 1 435 001 Active 0.88 Escherichia phage Rac⁃SA53 KU052037.1
MG1655 1 639 043 1 665 206 Active 0.83 Enterobacteria phage SfV U82619.2
MG1655 554 781 588 569 Ambiguous 0.66 Enterobacteria phage HK629 JQ182735.1
MG1655 1 196 209 1 211 578 Ambiguous 0.78 Shigella phage SfIV KC814930.1
MG1655 2 985 735 3 002 152 Ambiguous 0.60 Stx2⁃converting phage Stx2a_WGPS2 AP012537.1
MG1655 4 529 542 4 550 977 Ambiguous 0.66 Stx2⁃converting phage 86 AB255436.1