Page 10 - 南京医科大学学报自然科学版
P. 10
第41卷第7期
·940 · 南 京 医 科 大 学 学 报 2021年7月
A M 1 2 B M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
1 000 bp 1 000 bp
700 bp
500 bp 700 bp
400 bp 500 bp
300 bp 400 bp
200 bp
300 bp
100 bp
200 bp
C
ATGAGTTTGACGTGGATGAAGTGACAGAAGGCCCTGT
CTTCGCCATGTGCATGGCTGGTGCCCATGATCAGGCA
ACATTGATCAAGTGGATCCAAGGCTTGCAGGAGACCA
ACCCAACCCTGGCACAGATCCCAGTGGTATTCCGCCT
GGCCAGGTCCACGGGGGAGCTCCTGACAACTTCAAGG
CTGGAGGGACAGTCCCCTCCCCACAGTCCTCCCAAGG
GGCCTGAGGAAGATGATGAGACCATTCAGGCAGAGCA
GCAGCAGGGGCAAAGTTGAGTCAGATAGAGCTGGCAT
GAGGAGAAAAGACTGGGCTGGTATGAAATAACATGGA
AAAAAAAAAAAAAAAAA
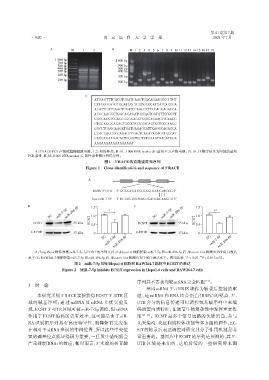
A:3′RACE⁃PCR产物琼脂糖凝胶电泳,1、2:阳性样品,M:DL 1 000 DNA marker;B:菌落PCR产物电泳,15、16、18测序结果为阳性的菌落
PCR条带,M:DL 1 000 DNA marker;C:阳性条带测序所得序列。
图1 3′RACE的克隆鉴定和序列
Figure 1 Clone identification and sequence of 3′RACE
A
ECSIT 3′UTR 5′ GCCCAGAUCCCCGUGGUCUUCCG 3′
hsa⁃miR⁃7⁃5P 3′ UGUUGUUUUAGUGAUCAGAAGGU 5′
B 1.5 C 1.5
miR⁃7⁃5P miR⁃296⁃5P * miR⁃7⁃5P miR⁃296⁃5P **
NC 1.0 NC 1.0
ECSIT 55 kDa ECSIT 0.5 ECSIT 55 kDa ECSIT 0.5
GAPDH 37 kDa 0 GAPDH 37 kDa 0
NC NC
miR⁃7⁃5P miR⁃296⁃5P miR⁃7⁃5P miR⁃296⁃5P
A:TargetScan软件预测miR⁃7⁃5p与ECSIT的序列互补;B:Hepa1⁃6细胞转染miR⁃7⁃5p和miR⁃296⁃5p后,Western blot检测ECSIT蛋白表达
水平;C:RAW264.7细胞转染miR⁃7⁃5p和miR⁃296⁃5p后,Western blot检测ECSIT蛋白表达水平。两组比较,P < 0.05,P < 0.01(n=3)。
**
*
图2 miR⁃7⁃5p抑制Hepa1⁃6细胞和RAW264.7细胞中ECSIT的表达
Figure 2 MiR⁃7⁃5p inhibits ECSIT expression in Hepa1⁃6 cells and RAW264.7 cells
[13]
序列并不要求与靶mRNA完全匹配 。
3 讨 论
基因 mRNA 3′⁃UTR 区域作为转录后控制的枢
本研究采用3′RACE实验获得ECSIT 3′⁃UTR区 纽,是 miRNA 和 RNA 结合蛋白(RBPs)的靶点,3′⁃
域的碱基序列;通过 miRNA 及 siRNA 干扰实验发 UTR 介导的信息传递可以调控氨基酸序列中未编
现,ECSIT 3′⁃UTR区域可被miR⁃7⁃5p调控,但siRNA 码的蛋白质特征,在调节生物复杂性中发挥重要作
作用于 ECSIT 编码区更有效率,这可能是由于 siR⁃ 用 [14-15] 。ECSIT 是多个信号通路的关键蛋白,参与
NA 识别靶序列具有高度特异性,而降解首先发生 先天免疫、炎症和线粒体功能等多方面的调节,EC⁃
在相对于 siRNA 来说的中间位置,所以这些中央位 SIT的转录后表达调控对研究其分子作用机制是非
置的碱基位点就显得极为重要,一旦发生错配就会 常重要的。基因库中ECSIT的序列是预测的,其3′⁃
严重抑制 RNAi 的效应,相对而言,3′末端的核苷酸 UTR 区域是未知的,这给后续的一些研究带来困