Page 62 - 南京医科大学学报自然科学版
P. 62
第44卷第2期
·200 · 南 京 医 科 大 学 学 报 2024年2月
A B Permutation testing
R2(0,0.998 3),Q2(0,0.7)
ALL
NC 1.0
20
( 19.08% ) 0 0.9 R2Y(cum)
t2
-20 0.8 Q2(cum)
-40 0.7
-30 -20 -10 0 10 000 0.25 0.50 0.75 1.00
t1(29.67%) Similarity(Y,Yperm )
C D R2(0,0.998 3),Q2(0,-0.03)
Permutation testing
[ 1 ] 25.1% ) ( 40 ALL 1.0
NC
20
Orthogonal T score -20 0 0 R2Y(cum)
0.5
Q2(cum)
-40
-10 -5 0 5 10 000 0.25 0.50 0.75 1.00
T score[1](23.2%) Similarity(Y,Yperm )
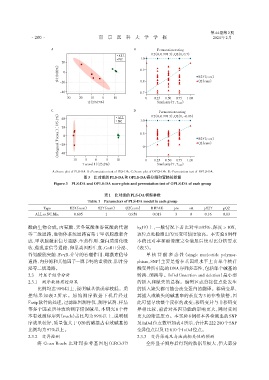
A:Score plot of PLS⁃DA. B:Permutation test of PLS⁃DA. C:Score plot of OPLS⁃DA. D:Permutation test of OPLS⁃DA.
图3 比对组的PLS⁃DA和OPLS⁃DA得分图和置换检验图
Figure 3 PLS⁃DA and OPLS⁃DA score plots and permutation test of OPLS⁃DA of each group
表1 比对组的PLS⁃DA模型参数
Table 1 Parameters of PLS⁃DA model in each group
Type R2X(cum) R2Y(cum) Q2(cum) RMSEE pre ort pR2Y pQ2
ALL.vs.NC.Mix 0.605 1 0.958 0.013 3 0 0.16 0.03
酸的生物合成,丙氨酸、天冬氨酸和谷氨酸的代谢 hg19)上,一般情况下若比对率≥95%、深度>10X,
等二级通路,生物体系统通路富集于甲状腺激素合 该位点处检测出的突变可信度较高。本实验8例样
成、甲状腺激素信号通路、生热作用、蛋白质消化吸 本的比对率和覆盖度完全满足后续对比分析要求
收、催乳素信号通路、卵巢类固醇生成、GnRH分泌、 (表3)。
谷氨酸能突触、FcγR⁃介导的吞噬作用、雌激素信号 单 核 苷 酸 多 态 性(single nucleotide polymor⁃
通路、内分泌和其他因子−调节钙的重吸收、胆汁分 phism,SNP)主要是指在基因组水平上由单个核苷
泌等二级通路。 酸变异所引起的DNA序列多态性,包括单个碱基的
2.3 外显子组学分析 转换、颠换等。InDel(insertion and deletion)是小型
2.3.1 测序数据质控结果 的插入和缺失的总称。编码区或剪接位点处发生
比例均达 90%以上,说明碱基错误率较低。质 的插入缺失都可能会改变蛋白的翻译。移码变异,
控结果如表 2 所示。原始测序数据下机后经过 其插入或缺失的碱基串的长度为 3 的非整数倍,因
Fastp软件的处理,过滤掉因测序仪、测序试剂、样品 此可能导致整个读框的改变;移码变异与非移码变
等多个因素所导致的测序错误碱基,本研究 8 个样 异相比较,前者对基因功能的影响更大,同时受到
本有效测序序列(reads)占比均为99%以上,说明测 更大的筛选压力。本实验8例样本各检测出的SNP
序效果较好,质量值大于 Q20 的碱基占有效碱基的 及InDel位点数量如表4所示,合计共222 280个SNP
比例均为97%以上。 变异位点以及12 610个InDel位点。
2.3.2 变异检测 2.3.3 变异筛选及与疾病相关性的预测
将 Clean Reads 比对到参考基因组(GRCh37/ 全外显子测序后得到的数据量极大,但大部分