Page 38 - 南京医科大学自然版
P. 38
第44卷第7期
·922 · 南 京 医 科 大 学 学 报 2024年7月
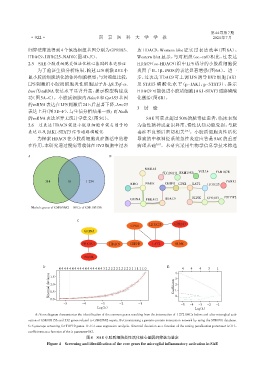
归算法筛选得到4个候选枢纽基因分别为GPR183、 达 HDAC9,Western blot 证实过表达效率(图 6A)。
HDAC9、LRRC25、NADK(图4D、E)。 Western blot 显示,与对照组(oe⁃ctrl)相比,过表达
2.5 SAE小胶质细胞炎性活化核心基因的表达验证 HDAC9(oe⁃HDAC9)组中 LPS 诱导的小胶质细胞促
为了验证生信分析结果,构建 LPS 刺激 BV2 小 炎因子IL⁃1β、iNOS 的表达显著增强(图6A)。进一
鼠小胶质细胞活化的体外细胞模型;与对照组比较, 步,过表达 HDAC9 可上调 LPS 诱导 BV2 细胞 JAK1
LPS 刺激组小胶质细胞炎性细胞因子 Il⁃1β、Tnf⁃α、 及 STAT3 磷 酸 化 水 平(p ⁃ JAK1、p ⁃ STAT3),提 示
Inos 的 mRNA 表达水平显著升高,提示模型构建成 HDAC9可能促进小胶质细胞JAK1⁃STAT3通路磷酸
功(图 5A~C)。小胶质细胞内 Hdac9 和 Gpr183 基因 化激活(图6B)。
的mRNA表达在LPS刺激后24 h后显著下降,Lrrc25
3 讨 论
表达上升(图5D~F),与生信分析结果一致;而Nadk
的mRNA表达差异无统计学意义(图5G)。 SAE 可累及超过 50%的脓毒症患者,临床表现
2.6 过表达HDAC9促进小胶质细胞中促炎因子的 为急性精神或意识异常、慢性认知功能受损,与脓
表达以及JAK1⁃STAT3信号通路磷酸化 毒症不良预后密切相关 [15] 。小胶质细胞炎性活化
为探索 HDAC9 在小胶质细胞炎症激活中的潜 导致的中枢神经系统急性炎症应答是 SAE 的重要
在作用,本研究通过慢病毒载体在BV2细胞中过表 病理基础 [16] 。本研究采用生物学信息学技术筛选
A B
MXRA8
VGLL4 FAM107B
SLC26A11 FAM198B
314 18 1 254 PANX1
KMO NADK CEBPB GPX1 LAT2 LRRC25
GRINA PRKAG1 HDAC9 BLNK GPR183 PSTPIP2
Module genes of GSE65682 DEGs of GSE103156
C
LRRC25 GPR183
GPX1
GRINA
PRKAG1 HDAC9 CEBPB LAT2 BLNK
NADK
D E
4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 3 2 2 2 2 2 2 2 2 2 2 1 1 1 1 1 0 4 4 4 4 3 1
Binomial deviance 1.0 Coeffcients 3 2 1
1.5
0.5
0.0 0
-5 -4 -3 -2 -1 -5 -4 -3 -2 -1
Log(λ) Log(λ)
A:Venn diagram demonstrates the identification of the common genes resulting from the intersection of 1 272 DEGs before and after microglial acti⁃
vation of GSE103156 and 332 genes related to GSE65682 sepsis. B:Constructing a protein⁃protein interaction network by using the STRING database.
C:Cytoscape screening for TOP10 genes. D,E:Lasso regression analysis. Binomial deviation as a function of the tuning penalization parameter λ(D),
coefficients as a function of the λ parameter(E).
图4 SAE小胶质细胞炎性活化核心基因的筛选与鉴定
Figure 4 Screening and identification of the core genes for microglial inflammatory activation in SAE