Page 96 - 南京医科大学学报自然科学版
P. 96
第42卷第7期
·992 · 南 京 医 科 大 学 学 报 2022年7月
A B C
*** 40
15 6 ** 30 ***
NSUN5相对表达量 10 5 *** NSUN6相对表达量 4 2 TET2相对表达量 20 ***
10
0 0 0
HK⁃2 786⁃O A498 HK⁃2 769⁃O A498 HK⁃2 769⁃O A498
D E F
***
6 3 *** 5 3 ***
DNMT3B相对表达量 4 2 *** NSUN5相对表达量 2 1 NSUN6相对表达量 2 1
0 0 0
HK⁃2 786⁃O A498 癌旁组织 癌组织 癌旁组织 癌组织
G H
3 * 3 *
TET2相对表达量 2 1 DNMT3B相对表达量 2
1
0 0
癌旁组织 癌组织 癌旁组织 癌组织
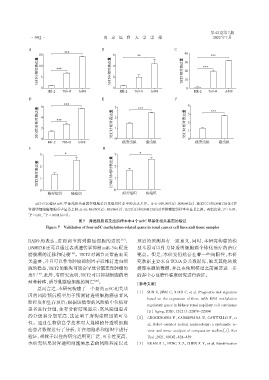
qRT⁃PCR 验证 m5C 甲基化相关基因在细胞系以及组织样本中的表达差异。A~D:NSUN5(A)、NSUN6(B)、TET2(C)和 DNMT3B(D)在
肾透明细胞癌细胞系中显著上调;E~H:NSUN5(E)、NSUN6(F)、TET2(G)和DNMT3B(H)在肿瘤组织样本中显著上调。两组比较,P < 0.05,
*
P < 0.01, P < 0.001(n=4)。
** ***
图7 肾癌细胞系及组织样本中4个m5C甲基化相关基因的验证
Figure 7 Validation of four m5C methylation⁃related genes in renal cancer cell lines and tissue samples
RAD9 的表达,进而调节前列腺癌细胞的进展 [17] , 预后的预测具有一定意义,同时,本研究构建的模
DNMT3B还可以通过表观遗传学抑制miR⁃34a促进 型基因可以作为肾透明细胞癌个体化治疗的治疗
膀胱癌的迁移和侵袭 [18] 。TET2 对调节正常造血至 靶点。但是,本研究仍然存在着一些局限性,本研
关重要,并且可以作为肿瘤抑制因子以维持造血细 究数据主要来自 TCGA 公共数据库,缺乏其他的数
胞的稳态,TET2的缺失可能会导致骨髓恶性肿瘤的 据库来源的数据,并且在使用模型之前需要进一步
发生 [19] ,此外,有研究表明,TET2可以抑制细胞的增 的多中心前瞻性临床研究进行验证。
[20]
殖和转移,诱导乳腺癌细胞的凋亡 。 [参考文献]
总而言之,本研究构建了一个新的m5C相关基
[1] SUN Z,JING C,XIAO C,et al. Prognostic risk signature
因的风险预后模型用于预测肾透明细胞癌患者风
based on the expression of three m6A RNA methylation
险程度和生存预后;根据该模型的风险值中位值对
regulatory genes in kidney renal papillary cell carcinoma
患者进行分组,生存分析结果显示:高风险组患者
[J]. Aging,2020,12(21):22078-22094
的分级和分期更高,这证明了所构建模型的可靠
[2] CROCEROSSA F,CARBONARA U,CANTIELLO F,et
性。通过生物信息学技术对大规模的肾透明细胞 al. Robot⁃assisted radical nephrectomy:a systematic re⁃
癌患者数据进行了分析,并在细胞系和组织中进行 view and meta ⁃ analysis of comparative studies[J]. Eur
验证,相较于以往的研究适用更广泛、可靠性更高, Urol,2021,80(4):428-439
本研究结果对肾透明细胞癌患者的风险程度以及 [3] RUAN B L,FENG X Z,CHEN X Y,et al. Identification