Page 53 - 南京医科大学学报自然科学版
P. 53
第42卷第8期 彭 鹏,解翠薇,王 军,等. 甲基苯丙胺的神经毒性:基于蛋白组学的靶向神经突触损伤研究[J].
2022年8月 南京医科大学学报(自然科学版),2022,42(08):1093-1099 ·1095 ·
75 min,B 液线性梯度 6%~38%;75~85 min,B 液线 光 4 ℃过夜,后加入荧光标记二抗及防荧光猝灭
性梯度 38%~100%;85~90 min,B液维持在100%。 DAPI 复合染料,激光共聚焦显微镜下拍照,计数。
质谱鉴定:样品经色谱分离后用 Q Exactive 质 每皿细胞随机拍摄9个视野,实验重复3次。
谱仪进行质谱分析。利用多肽和多肽碎片的质量 1.3 统计学方法
电荷比采集,每次全扫描后采集 10 个碎片图谱 实验数据统计分析处理,使用SPSS20.0软件进
(MS2 scan),仪 器 条 件 设 置 如 下 :MS2 Activation 行统计学分析,多组定量资料比较采用单因素方差
Type 为 HCD,Isolation window 为 2 m/z,二级质谱分 分析(one⁃way ANOVA)检验,多组数据间两两比较
辨率 35 000,Microscans 为 1,二级 Maximum IT 为 选用Dunnett⁃t检验,两组定量数据比较用Student’s
45 ms,Normalized Collision Energy 为 30 eV。 t 检验,P < 0.05 为差异有统计学意义,绘图使用
蛋白质定性和定量分析:通过 Proteome Discov⁃ GraphPad Prism 7.0。
erer 2.1软件内置工具提交到 MASCOT2.6 服务器进
2 结 果
行数据库检索;通过Proteome Discoverer 2.1 将MAS⁃
COT 服务器上形成的查库文件(.dat 文件)传回软 2.1 同位素标记相对和绝对定量(iTRAQ)分析
件,根据FDR<0.01 的标准对数据进行筛选,获得高 同位素标记相对和绝对定量(isobaric tag for
度可信的定性结果;使用数据库 Uniprot_RattusNor⁃ relative and absolute quantitation,iTRAQ)是一种体
vegicus_36080_20180123,针对质谱分析原始文件用 外同位素标记的蛋白质定性定量技术。为观察
软件Mascot2.6和Proteome Discoverer2.1进行查库鉴 METH 对 原 代 神 经 元 蛋 白 质 的 表 达 改 变 ,通 过
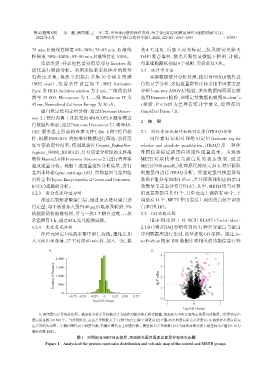
定及定量分析。根据上述定量定性分析结果,进行 METH(900 μmol/L)处理原代神经元24 h,然后提取
基因本体论(gene ontology,GO)、京都基因与基因组 细胞蛋白进行 iTRAQ 分析。所鉴定蛋白按差异倍
百科全书(Kyoto Encyclopedia of Genes and Genomes, 数和 P 值分布如图 1 所示,差异倍数和相应的蛋白
KEGG)通路的分析。 质数量呈正态分布(图 1A),其中,METH 组与对照
1.2.3 蛋白免疫印迹分析 组比差异蛋白共51个,其中表达上调的有40个,下
神经元裂解提取蛋白后,通过BCA法对蛋白进 调的有11个,METH作用表达上调的蛋白较下调蛋
行定量,每个泳道加入蛋白40 μg后电泳及转膜,5% 白多(图1B)。
的脱脂奶粉溶液封闭,后与一抗4 ℃孵育过夜,二抗 2.2 GO功能注释
常温孵育1 h,通过ECL发光检测成像。 用 序 列 比 对 工 具 NCBI BLAST +(ncbi ⁃ blast ⁃
1.2.4 免疫荧光分析 2.3.0+)将 iTRAQ 分析得到的 51 种差异蛋白与蛋白
神经元固定(4%的多聚甲醛),清洗,透化后加 序列数据库进行比对,批量提取GO 注释。通过 In⁃
入10%山羊血清,37 ℃封闭60 min后,加入一抗,避 terProScan 搜索 EBI 数据库将相关的功能信息注释
A B
4
2 000 3
Number of Proteins 1 500 ⁃lg P value 2
1 000
500
0 1
-0.75 -0.50 -0.25 0 0.25 0.50 0.75
0
log2 fold change
-1 0 1
log2 fold change
A:两组蛋白差异的分布图。横坐标为将差异倍数以 2 为底作对数变换后所得数值,纵坐标为本次实验鉴定的蛋白质数量。结果鉴定出
蛋白质总数为6 619个。与对照组比,表达差异倍数大于1.2倍(包含上调/下调蛋白)且P值<0.05的蛋白质为差异蛋白;B:两组样本蛋白质表
达差异的火山图。左侧红圈代表下调蛋白数,右侧红圈代表上调蛋白数。横坐标为差异倍数(以2为底的对数变换);纵坐标为P值(以10为
底的对数变换)。
图1 对照组与METH比较后,两组样本蛋白质表达差异分布和火山图
Figure 1 Analysis of the protein expression distribution and volcanic map of the control and METH groups