Page 39 - 南京医科大学自然版
P. 39
第44卷第6期 刘嘉浩,钱海声,高 欣,等. 基于生物信息学分析干性相关基因TCEAL7作为胃癌预后
2024年6月 标志物的研究[J]. 南京医科大学学报(自然科学版),2024,44(6):769-780 ·775 ·
A 16 ** ** ** ** ** ** ** ** ** ** ** ** ** ** * ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** **
14 ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** ** **
12
10
8 6
Expression 4 2 Group
Tumor
-2 0 Normal
-4
-6
-8
-10
N=1 151)
N=167)
N=1 151) N=1 151) N=23) N=292) N=13) N=396) N=664) N=873) N=167) N=167) N=345) N=355) N=152) N=209) N=44) N=167) N=396) N=159) N=167) N=558) N=28) N=337) N=10) N=88) N=169) N=165) N=78) N=199) N=199) N=3) N=127) (T=36, N=9)
(T=418,
(T=365,
(T=380,
(T=883,
(T=503,
(T=147,
(T=513,
(T=595,
(T=1 092,
(T=30,
(T=77,
(T=529,
(T=414,
(T=662,
(T=153, (T=509, (T=179, (T=303, (T=181, (T=288, (T=288, (T=495, (T=515, (T=498, (T=120, (T=85, (T=407, (T=92, (T=178, UCS (T=57, (T=24, (T=177, (T=66,
CHOL
PCPG
OV
COAD
GBM LGG UCEC BRCA CESC LUAD ESCA STES KIRP KIPAN COADREAD PRAD STAD HNSC KIRC LUSC LIHC WT SJCN BKCA THCA READ PAAD TGCT ALL LAML ACC KICH
GBMLGG
B Cancer code P Hazard ratio(95%CI)
TCGA⁃STAD(N=372) 1.7e⁃4 1.22(1.10-1.36)
TCGA⁃STES(N=547) 7.0e⁃4 1.16(1.07-1.27)
TCGA⁃GBMLGG(N=619) 1.7e⁃3 1.32(1.11-1.56)
TCGA⁃BLCA(N=398) 0.01 1.12(1.02-1.22)
TCGA⁃SKCM⁃P(N=80) 0.02 1.40(1.04-1.88)
TCGA⁃ACC(N=77) 0.02 1.51(1.06-2.15)
TCGA⁃KIRP(N=276) 0.03 1.17(1.02-1.35)
TCGA⁃KIPAN(N=854) 0.03 1.09(1.01-1.18)
TCGA⁃KICH(N=64) 0.08 1.82(0.93-3.53)
TCGA⁃ESCA(N=175) 0.20 1.12(0.94-1.32)
TCGA⁃THCA(N=500) 0.22 1.30(0.86-1.97)
TCGA⁃COAD(N=278) 0.29 1.10(0.93-1.30)
TCGA⁃LUSC(N=468) 0.37 1.05(0.95-1.16)
TCGA⁃LGG(N=474) 0.41 1.09(0.88-1.35)
TCGA⁃GBM(N=144) 0.41 1.11(0.87-1.42)
TCGA⁃COADREAD(N=368) 0.42 1.07(0.91-1.25)
TCGA⁃LIHC(N=337) 0.42 1.06(0.92-1.22)
TCGA⁃UVM(N=66) 0.45 1.08(0.88-1.32)
TCGA⁃OV(N=406) 0.47 1.03(0.95-1.13)
TCGA⁃HNSC(N=506) 0.56 1.03(0.94-1.12)
TCGA⁃THYM(N=115) 0.58 1.08(0.81-1.44)
TCGA⁃MESO(N=84) 0.59 1.03(0.91-1.17)
TCGA⁃DLBC(N=44) 0.81 1.07(0.61-1.88)
TCGA⁃BRCA(N=1 044) 0.87 1.01(0.90-1.14)
TCGA⁃LUAD(N=490) 0.91 1.01(0.89-1.15)
TCGA⁃SKCM⁃M(N=340) 0.95 1.00(0.92-1.09)
TCGA⁃CESC(N=272) 0.96 1.00(0.86-1.17)
TCGA⁃READ(N=90) 0.98 1.01(0.65-1.55)
TCGA⁃KIRC(N=514) 0.03 0.88(0.79-0.99)
TCGA⁃PRAD(N=492) 0.15 0.69(0.41-1.15)
TCGA⁃LAML(N=19) 0.15 0.69(0.40-1.18)
TCGA⁃CHOL(N=33) 0.24 0.77(0.50-1.19)
TCGA⁃UCS(N=55) 0.39 0.94(0.82-1.08)
TCGA⁃PCPG(N=170) 0.64 0.89(0.53-1.48)
TCGA⁃PAAD(N=172) 0.82 0.98(0.84-1.15)
TCGA⁃SKCM(N=420) 0.84 0.99(0.92-1.07)
TCGA⁃SARC(N=253) 0.89 0.99(0.92-1.07)
TCGA⁃TGCT(N=127) 0.91 0.97(0.56-1.67)
TCGA⁃UCEC(N=166) 0.97 1.00(0.91-1.23)
-1.2 -1.0 -0.8 -0.6 -0.4 -0.2 0 0.2 0.4 0.6 0.8 1.0 1.2 1.4 1.6 1.8
log2 [Hazard ratio(95%CI)]
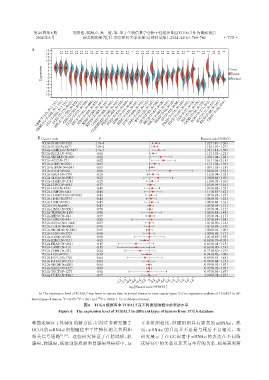
A:The expression level of TCEAL7 was lower in tumors than in normal tissues in most cancer types. B:Cox regression analysis of TCEAL7 in dif⁃
**
*
ferent types of tumors. P < 0.05,P < 0.01 and **** P < 0.000 1. CI:Cofidence interval.
图6 TCGA数据库中TCEAL7在不同类型肿瘤中的表达水平
Figure 6 The expression level of TCEAL7 in different types of tumors from TCGA database
种描述癌症干性特征的新方法,后续许多研究基于 正常组织相比,肿瘤组织具有更高的 mRNAsi。然
OCLR 的 mRNAsi 识别癌症中干性特征相关基因和 而,mRNAsi 的升高并不总是与预后不良相关。本
相关信号通路 [8-10] 。这些研究验证了在膀胱癌、乳 研究展示了在 GC 患者中 mRNAsi 的表达在不同临
腺癌、肺腺癌、低级别胶质瘤和胃腺癌等癌症中,与 床特征中的关系以及其与生存的关系,结果表明高